Pandemie / Auf der Spur des Virus: So kommen die Forscher des LNS von der Probe zur genauen Corona-Variante

Die Coronavirus-Proben werden sorgfältig in kleine Reagenzgläser gefüllt
Noch ansteckender, noch gefährlicher: Forscher warnen seit Wochen vor neuen Coronavirus-Varianten, die das Infektionsgeschehen wieder ankurbeln können. In Luxemburg untersuchen die Spezialisten des Nationalen Gesundheitslabors (LNS), welche Varianten des Virus im Großherzogtum präsent sind. Wir haben nachgefragt, wie man eigentlich vom Coronatest zur erfolgreichen Identifizierung des Virus kommt.
Das Coronavirus hat Luxemburg auch nach einem Jahr Pandemie noch fest im Griff. Doch mittlerweile hat sich eine ganz bestimmte Variante im Großherzogtum durchgesetzt: die sogenannte „britische“ B.1.1.7-Mutation. Sie soll ansteckender sein als die bisher vorherrschende Variante. Festzustellen, welche Mutationen des Coronavirus in Luxemburg ihr Unwesen treiben, ist die Aufgabe der Forscher des „Laboratoire national de santé“. Zum Team gehören unter anderem die Labortechnikerin Jessica Tapp, die Bioinformatikerin Dr. Anke Wienecke-Baldacchino und der Virologe Dr. Trung Nguyen Nguyen.
Bei etwa 400 Proben wird pro Woche in Luxemburg untersucht, um welche Variante des Virus es sich genau handelt. „Wir wollen diese Woche versuchen, es auf 600 Proben hochzuschrauben“, sagt Jessica Tapp. „Das Endziel sind etwa 800 pro Woche.“ Ob sie das schaffen, hänge zum einen davon ab, ob sie genug Material erhalten. Durch die weltweit gestiegene Nachfrage seien bestimmte Verbrauchsmaterialien immer wieder vergriffen. Andererseits ist das LNS auf der Suche nach den passenden Fachkräften. „Wir rekrutieren wie verrückt“, erzählt Tapp. Doch die passenden Profile zu finden, sei gar nicht so einfach. Auch weil, wie beim Material, überall nach diesen Spezialisten gesucht wird. Bei der Besichtigung der Labore des LNS fällt auf, dass die meisten der Forscher in ihren weißen Kitteln junge Frauen sind. Sie sitzen und arbeiten mit hoch spezialisierten Maschinen. Einige Geräte kosten mehrere 100.000 Euro. Vom Einreichen der Probe bis zur endgültigen Identifikation der Virus-Variante dauert es fünf bis sieben Tage.
Das LNS untersucht Coronavirus-Proben, die beim hauseigenen Drive-in, in den Krankenhäusern und dem „Centre de consultation Covid“ auf Kirchberg entnommen wurden. Priorität hätten die am Drive-in entnommenen Proben sowie die der Krankenhäuser. „Und natürlich, wenn sich irgendwo ein größeres Cluster bildet“, erklärt Virologe Dr. Trung Nguyen Nguyen. Um eine Repräsentativität der Daten zu garantieren, würden nun auch Proben aus den Privatlaboren das LNS zur DNA-Analyse erreichen. Die haben aber meist einige Tage Verspätung. Die Proben kommen über ein internes Postsystem auf dem dritten Stock des Gebäudes an. Ist eine Lieferung unterwegs, werden die Techniker über E-Mail informiert – und zur Sicherheit gibt es auch ein rotes Licht an der Tür, falls noch Proben abzuholen sind.

Die Röhrchen mit den entnommenen Abstrichen sind außen mit einem Strichcode versehen. Von welchem Patienten die Probe also genau stammt, sieht der Techniker nicht. Diese Daten sind im internen Laborinformationssystem gespeichert. Am Anfang der Krise waren Privatlabore, Krankenhäuser und das LNS noch nicht so aufeinander abgestimmt. Damals mussten die Daten nochmals per Hand im System des LNS eingegeben werden. Laut dem Virologen Nguyen Nguyen ist das heute einfacher: „Wir bekommen eine Excel-Tabelle und schon ist alles in unserem System.“
Der ganz normale PCR-Test
Für die Probe geht es zunächst einmal durch einen „normalen“ PCR-Test. Denn erst muss geklärt werden: Ist das Virus überhaupt präsent und ist die Virenlast hoch genug für eine DNA-Sequenzierung? Dazu entnimmt eine Mitarbeiterin sorgsam die Wattestäbchen mit den Abstrichen. Die Flüssigkeit in den Röhrchen hat in Zwischenzeit die so entnommenen Proben aufgenommen. Die Stäbchen selbst landen im Müll. Damit sie sich nicht ansteckt oder die Proben kontaminiert werden, sitzt die Mitarbeiterin mit Maske an einer besonderen Abzugshaube und vor einem Glasschutz. Sind die Proben alle vorbereitet, werden sie in jeweils vier Reihen in die erste Maschine eingeführt.

„Bei einem Abstrich entnimmt man nicht nur Viren, sondern auch alles mögliche andere“, erklärt Dr. Anke Wienecke-Baldacchino. Bakterien oder Hautzellen interessieren die Forscher nicht. Beim zweiten Arbeitsschritt geht es also darum, die Probe von allen möglichen Verunreinigungen zu befreien, sodass für den PCR-Test rein das Virus übrigbleibt. Diese Aufgabe wird von einer Maschine gestemmt. Das LNS besitzt aktuell drei solcher spezialisierten Geräte. Das neueste ist derzeit noch nicht am Start, soll aber in Kürze den Dienst antreten und könnte in etwa drei Stunden statt knapp 94 Proben 380 gleichzeitig vorbereiten. Danach werden die Proben automatisch verschlossen. „Wir berühren sie nicht, um eine Kontaminierung zu vermeiden.“

Von der wissenschaftlichen „Waschmaschine“ wandert die Probe nun zur „Polymerase-Kettenreaktion“, allgemein besser bekannt als PCR-Test. Tatsächlich ist es auf dem Weg zur DNA-Analyse aber nur der erste PCR-Test, den die Probe durchlaufen wird. Bei diesem wird das reine Probenmaterial mit Fluoreszenzsonden vermischt und in eine Art Kombination aus Ofen und Kühlschrank gesteckt. Das Gerät heißt Thermocycler und wird die Probe nun abwechselnd unterschiedlichen Temperaturen aussetzen. So wird das Erbmaterial verdoppelt. Ist Erbmaterial des Virus, die Ribonukleinsäure (RNA), vorhanden, setzen die Sonden sich da ab und fangen an zu leuchten. Das Gerät erkennt dies und der blaue Punkt am Bildschirm, der die Probe repräsentiert, wird rot: Der Mensch, dem diese Probe gehört, trägt das Virus in sich. Abhängig davon, wie schnell der Thermocycler die positive Probe entdeckt – also nach wie vielen Temperaturzyklen – gibt es einen CT-Wert. CT steht für Cycle Threshold, zu Deutsch Schwellenwert-Zyklus, also wie viele Zyklen durchlaufen werden. Liegt dieser sehr niedrig – schlägt das Gerät also schnell an –, ist viel Virus enthalten. Dieser Infizierte ist also deutlich ansteckender. Proben von Menschen, die erst am Anfang oder aber am Ende der Infektion stehen, haben häufig deutlich höhere CT-Werte. Geht es nur darum, zu bestimmen, ob jemand positiv ist, ist die Reise hier zu Ende.

Von der RNA zur DNA
Für die DNA-Analyse braucht es einen hohen CT-Wert: „Sonst finden wir nicht genügend Material“, erklärt Labortechnikerin Tapp. Sie und ihr Team übernehmen die besten Proben aus dem ersten PCR-Test. Mindestanforderung sind CT-Werte unter 35. Sie sind dafür zuständig, dass aus der RNA des Virus Desoxyribonukleinsäure, also die bekannte DNA, wird. Dies nennt man in der Fachsprache „Reverse Transcription“. Der Unterschied zwischen DNA und RNA ist der Aufbau des Erbmaterials. Die DNA ist eine „Doppelhelix“, sieht also aus wie eine gewundene Leiter. Die RNA windet sich zwar auch, hat aber nur eine Helix – gleicht also eher einer in der Hälfte abgesägten Leiter.

Das Basisgenom des Virus ist 30.000 Basispaare (bp) – also Stufen – lang. „Das können wir nicht an einem Stück durchsequenzieren“, sagt Tapp. Deswegen „zerschneiden“ sie und ihre Kollegen das Genom in „mundgerechte“ Stücke für die nächste PCR-Maschine. Die Leiter wird auf Stücke von 900 Stufen gekürzt und es werden zusätzliche „Bausteine“ in das Mini-Röhrchen gekippt. Dann wandert die Probe wieder in den Thermocycler für knapp sechseinhalb Stunden. Diesmal für die Amplifikation des Virus-Erbguts. „Das tut sich über Nacht“, sagt Tapp. Im LNS werden so zwei Plaketten mit je 96 Proben an vier Tagen der Woche verarbeitet.
Nach dem Amplicon-PCR muss die Probe noch von den überflüssigen Bausteinen gereinigt werden. Dazu setzen die Techniker den kleinen Röhrchen eine bräunliche Substanz bei, dort enthalten sind winzige Metallkügelchen. Die Platte wird danach auf einen speziellen magnetischen Untersetzer gestellt. Der zieht die Kügelchen nach unten, die so das Erbgut einfangen. Der Rest der Probe ist überflüssig und kann entsorgt werden. Bisher machen diese Arbeitsschritte zwei Labortechniker ganz von Hand. Doch bald soll auch eine spezialisierte Maschine für Unterstützung sorgen.

Was jetzt in den Behältern zurückbleibt, sind 900 bp große Stückchen pure Virus-DNA – noch etwas zu große Häppchen für das Sequenziergerät. Sie müssen weiter in 150 bp lange Ketten zerkleinert werden, dies wird „Library Prepartion“ genannt. Diese Etappe wird ebenfalls von zwei Labortechnikern manuell ausgeführt. Bei der Sequenzierung stellen die Forscher nun fest, aus welchen „Bausteinen“ die jeweiligen Stufen der DNA gefertigt sind. Das können die Basen Adenin (A), Guanin (G), Cytosin (C) und Thymin (T) sein. Sie sind dabei etwas eigenwillig: Sie treten nur in bestimmten Gruppierungen auf. Also A und T arbeiten immer zusammen und G hält sich strikt an C. Im Sequenziergerät wird ein Teil der Leiter von der anderen getrennt und dann beobachtet, wo welche Bausteine wieder andocken. Passiert das, macht das Gerät ein Foto mit dem glücklichen Paar, das sich gefunden hat. Auf einem Mini-Chip wird gespeichert, wann in den 150 bp langen Strängen „Fotos“ geschossen werden – und wer dort zu erkennen ist.
Mutation gesucht
Was die Maschine am Ende ausspuckt, kommt bei Dr. Anke Wienecke-Baldacchino im Büro an. Das sind rund 192 Proben gleichzeitig. Ihre Aufgabe ist es nun, das Genom aus den kurzen Sequenzen wieder zusammenzusetzen. Dazu muss sie diese erst mal säubern, also Sequenzen, die etwa nicht hochwertig genug für eine Analyse sind, herausnehmen oder fehlerhafte Stücke abschneiden. Auch entfernt ein Programm über Nacht – acht Stunden sind nötig –alle noch verbliebenen Überreste von menschlicher DNA.
Dann werden die Sequenzen mit dem Referenzgenom, also einer Art genetischem Passbild des Virus, verglichen. Wo dem Computer Abwandlungen auffallen, also etwa ein G anstelle eines T auftaucht, wird die Sequenz mit der zugeordneten Farbe des Bausteins eingefärbt. Taucht die Veränderung des Genoms nur in einer Sequenz auf, handelt es sich in der Regel um einen Sequenzierfehler. Ist die Veränderung aber bei allen Sequenzen oder einer großen Mehrheit zu finden, ist eine Mutation aufgetreten.
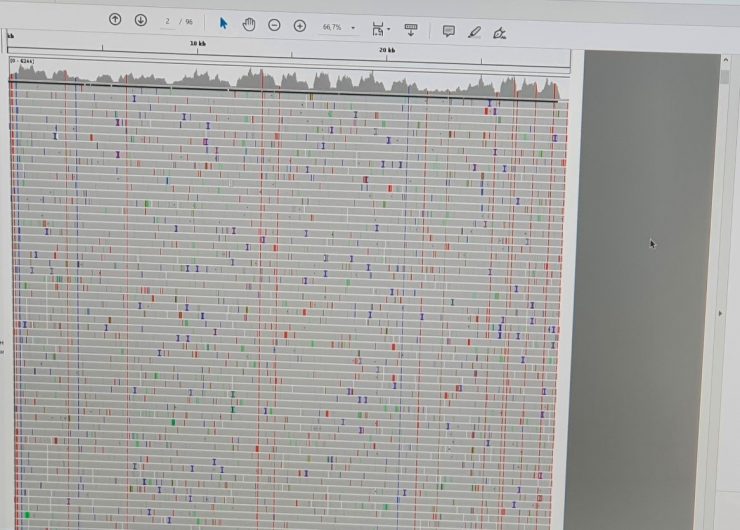
Um herauszufinden, wie die genaue DNA einer positiven Probe aussieht, „kollabiert“ Wienecke-Baldacchino die einzelnen Sequenzen übereinander und macht eine „Konsensussequenz“, also eine Art Fahndungsbild des Virus-Übeltäters. Das wird dann in „Pangolin“ eingespeist, eine Datenbank, wo die „Fotos“, also DNA-Sequenzen, aller Coronavirus-Varianten liegen. Jede hat nämlich eine oder mehrere bestimmte Mutationen. Passt das Fahndungsbild auf ein bestimmtes Foto, sagt der PC der Wissenschaftlerin innerhalb von Sekunden: „Das ist B 1.1.7“. Von den ursprünglichen Daten, die aus dem Sequenziergerät kommen, bis zu diesem Fahndungserfolg dauert es etwa vier Stunden. Die komplette Analyse dauert acht Stunden und kann auch mal über Nacht laufen.

Dass man von einer „britischen“ oder „südafrikanischen“ Variante spricht, ist nicht ganz richtig, erklärt die Forscherin. „Der Name kommt von dem Land, das die Sequenzen als Erstes in die Datenbank einfügt. Das bedeutet nicht zwangsläufig, dass die Mutation auch dort entstanden ist. Nur dass man sie dort zuerst entdeckt hat.“ Luxemburg liege beim Sequenzieren weltweit unter den Spitzenreitern. Ausgeschlossen, dass es irgendwann eine „luxemburgische“ Variante gibt, ist es nicht. Bisher sei das aber nicht der Fall gewesen.
Mutationen entstehen spontan und zufällig. „Auch wenn es so aussieht, das Virus weiß nicht, was es tut“, sagt Wienecke-Baldacchino. Was sich durchsetzt, sind die Varianten, die sich durch ihre Mutationen einen Vorteil verschaffen konnten. Andere sterben mit der Zeit aus. Wie etwa das tatsächliche Original SARS-CoV-19-Genom, der sogenannte „Wuhan“-Strang.
Interessante Proben speichert sich die Bioinformatikerin auch ab. Etwa, wenn bei einem Genom, das als „B.1.1.7“ klassiert wurde, auch eine Mutation vorhanden ist, die normalerweise bei einer anderen Variante vorkommt. „Dabei könnte es sich etwa um eine doppelte Infektion handeln. Der Patient könnte sich mit mehreren Coronavirus-Varianten angesteckt haben.“
Positiv getestete Personen bekommen übrigens nicht mitgeteilt, mit welcher Virus-Variante sie sich angesteckt haben, bestätigt das Gesundheitsministerium gegenüber dem Tageblatt. Das LNS ist hier nicht zuständig, es gibt die Daten nur an die Gesundheitsinspektion weiter. „Auch wenn die Leute hier anrufen, wir können ihnen nicht weiterhelfen“, sagt Wienecke-Baldacchino. Sie habe aber Verständnis dafür und empfindet es auch als sinnvoll, wenn das den Infizierten gesagt würde. „Besonders wenn es sich um die britische Variante handelt. Wissenschaftler gehen davon aus, dass Infizierte mit dieser Variante länger ansteckend sind.“ Das Gesundheitsministerium bestätigt gegenüber dem Tageblatt, dass Personen, die mit einer Variante des Coronavirus infiziert sind, nicht länger als üblich in Isolierung bleiben müssen. „Das sieht das Gesetz nicht vor.“
- Luxemburger Ermittler finden hunderte Kilo Drogen in importierter Zerkleinerungsmaschine - 7. Februar 2025.
- Die Kandidaten des Luxembourg Song Contest im Überblick - 26. Januar 2025.
- Musik, Spezialgäste und eine Abstimmung: Das erwartet die Zuschauer während der Show - 25. Januar 2025.



 Headlines
Headlines









 Umfrage
Umfrage
 Facebook
Facebook  Twitter
Twitter  Instagram
Instagram  LinkedIn
LinkedIn
Faszinierender Artikel. Komplizierte Wissenschaft, für den Laien in verständlichen Worten beschrieben. Danke!
Super und gut verständlich